Post by German Grekhov on Aug 9th, 2013 at 10:36am
Здравствуйте. Извините за долгий ответ.
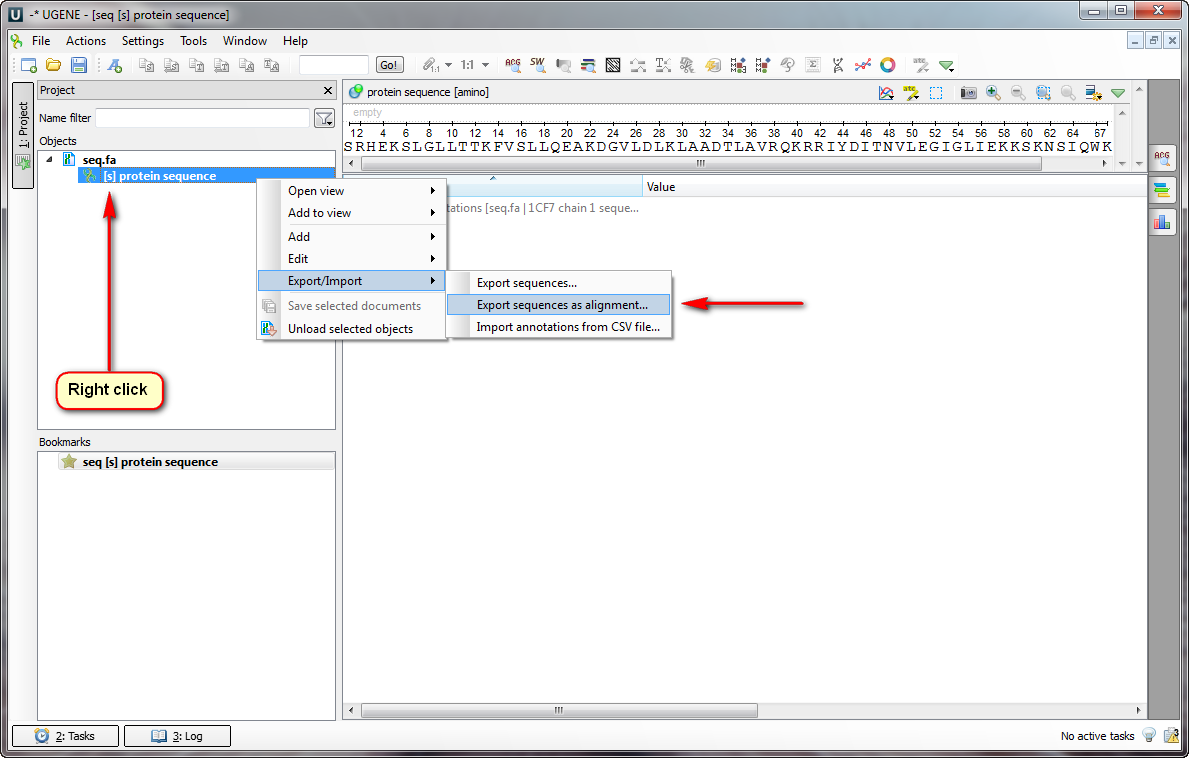
Чтобы получить HMMER-профиль из одной белковой последовательности, первым делом нужно превратить (экспортировать) эту последовательность в выравнивание. Посмотрите, пожалуйста, первую приложенную картинку.
1) Найдите вашу последовательность в проекте и правой кнопкой мыши вызовите ее контекстное меню.
2) Перейдите по пунктам Import/Export -> Export sequences as alignment.
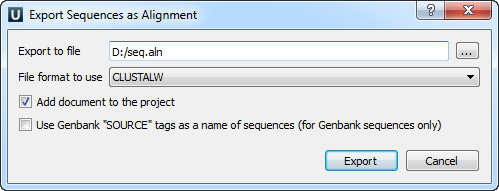
В результате вы увидите диалог экспортирования последовательности (рисунок 2), в котором нужно задать путь до нового файла, в который будет сохранено выравнивание. После нажатия кнопки Export новое выравнивание автоматически откроется в UGENE.
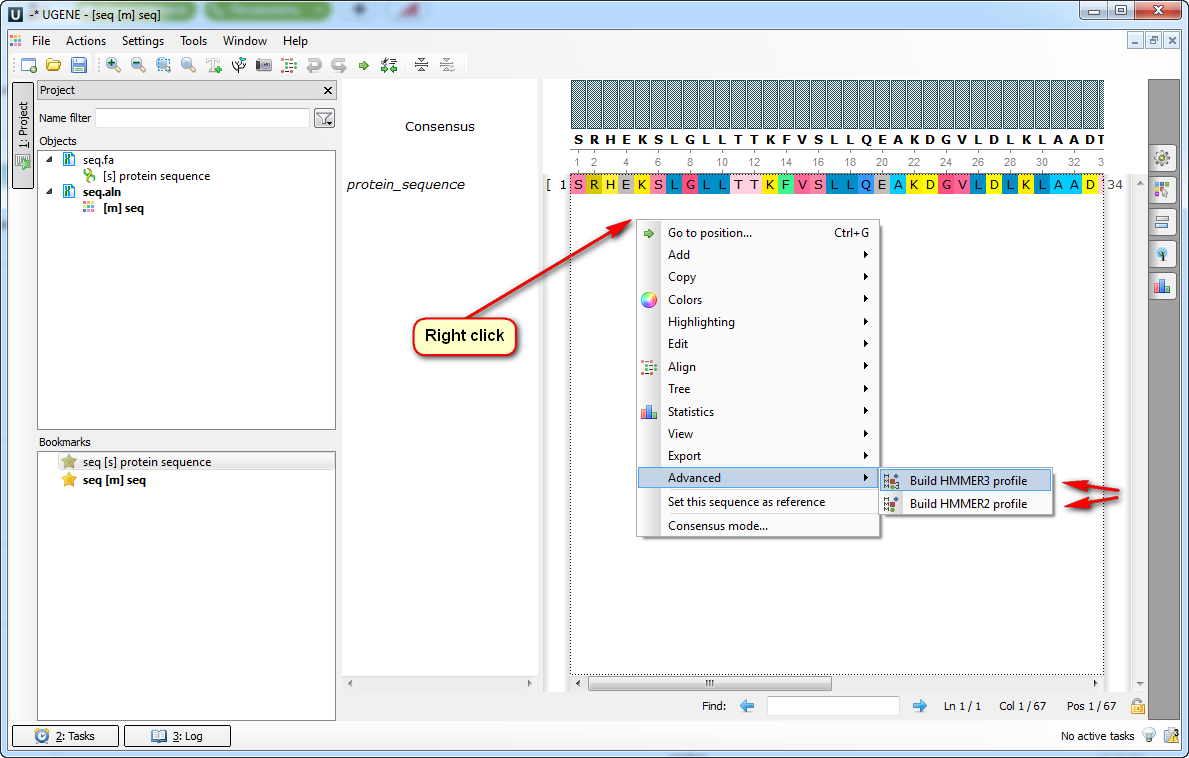
В редакторе множественных выравниваний (рисунок 3) правой кнопкой мыши вызовите контекстное меню и перейдите по пунктам Advanced -> Build HMMER3 profile (или HMMER2).
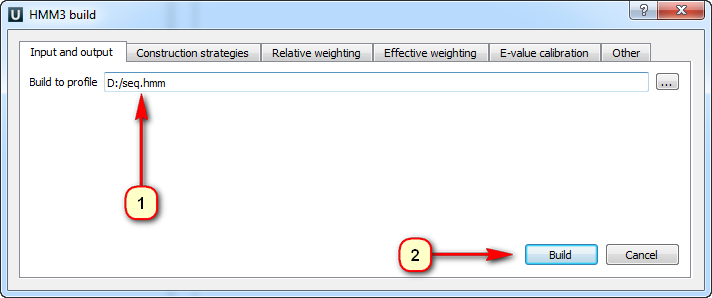
В полученном диалоге (рисунок 4) вы можете выбрать путь до нового файла с HMMER-профилем, а также настроить множество параметров построения, которые находятся во вкладках диалога.
Если возникнут дальнейшие вопросы, пожалуйста, задавайте их. Теперь постараемся ответить как можно быстрее.
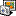 1_010.png (112 KB | )
1_010.png (112 KB | )
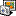 2_009.png (14 KB | )
2_009.png (14 KB | )
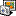 3_003.png (82 KB | )
3_003.png (82 KB | )
Чтобы получить HMMER-профиль из одной белковой последовательности, первым делом нужно превратить (экспортировать) эту последовательность в выравнивание. Посмотрите, пожалуйста, первую приложенную картинку.
1) Найдите вашу последовательность в проекте и правой кнопкой мыши вызовите ее контекстное меню.
2) Перейдите по пунктам Import/Export -> Export sequences as alignment.
В результате вы увидите диалог экспортирования последовательности (рисунок 2), в котором нужно задать путь до нового файла, в который будет сохранено выравнивание. После нажатия кнопки Export новое выравнивание автоматически откроется в UGENE.
В редакторе множественных выравниваний (рисунок 3) правой кнопкой мыши вызовите контекстное меню и перейдите по пунктам Advanced -> Build HMMER3 profile (или HMMER2).
В полученном диалоге (рисунок 4) вы можете выбрать путь до нового файла с HMMER-профилем, а также настроить множество параметров построения, которые находятся во вкладках диалога.
Если возникнут дальнейшие вопросы, пожалуйста, задавайте их. Теперь постараемся ответить как можно быстрее.
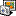 1_010.png (112 KB | )
1_010.png (112 KB | )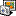 2_009.png (14 KB | )
2_009.png (14 KB | )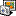 3_003.png (82 KB | )
3_003.png (82 KB | )