Post by Ilya Flyamer on Jul 23rd, 2014 at 6:24pm
Юрий,
ясно, спасибо, надо будет попробовать, когда выйдет 1.14. Кажется, там намечается много полезного!
Ольга,
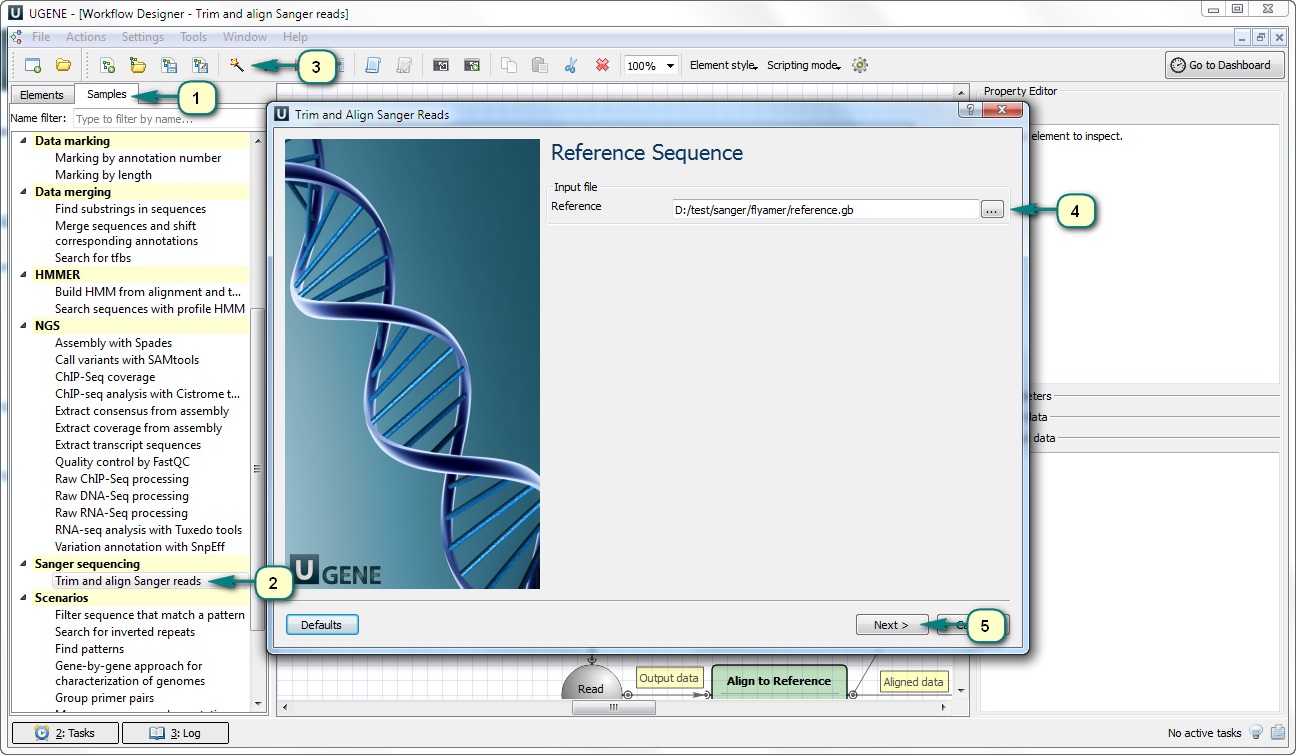
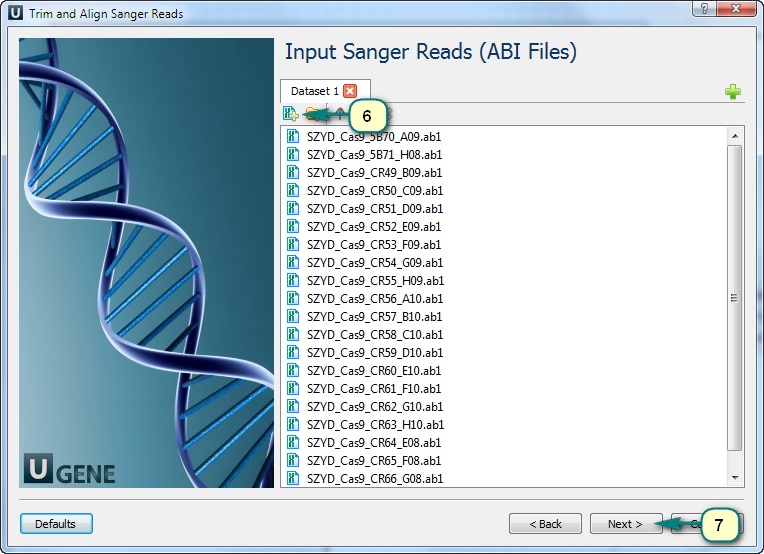
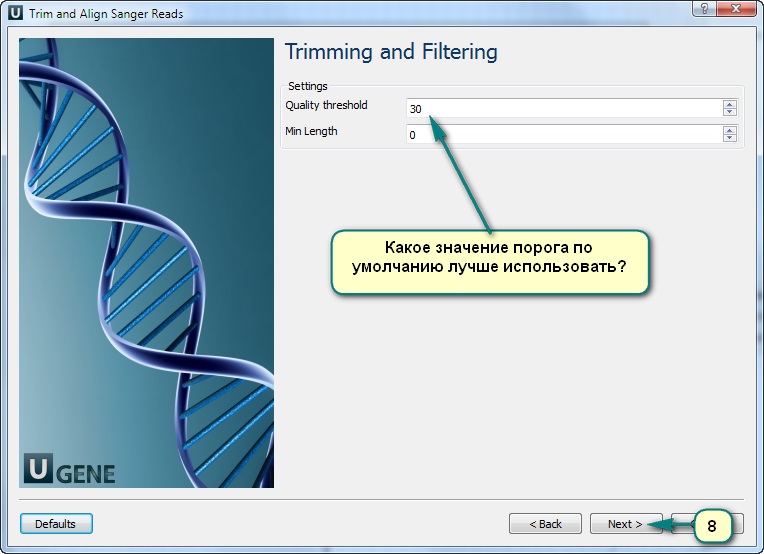
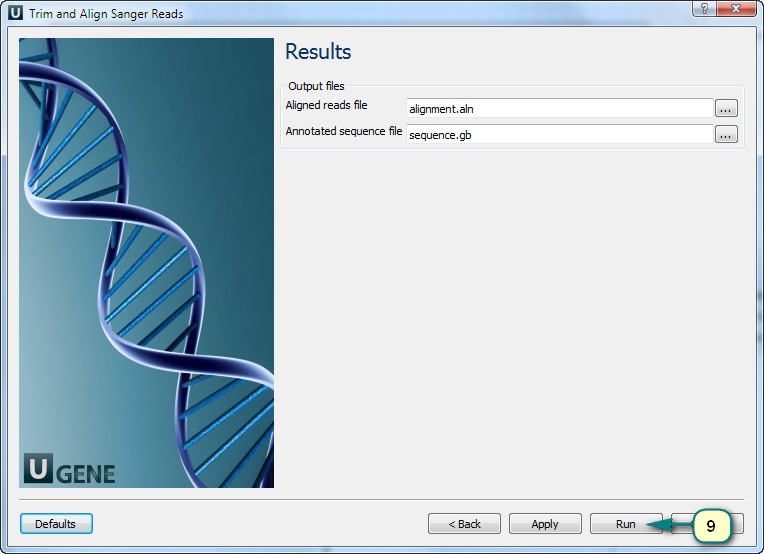
вот архив с ab1 файлами, которые я как раз использую: https://yadi.sk/d/EWIHJjgSXBRST
А во вложении последовательность плазмиды, которую я проверяю.
 https://forum.ugene.net/forum/YaBB.pl?action=downloadfile;file=pX330a_Zhang_vector_by_Addgene.gb (16 KB | )
https://forum.ugene.net/forum/YaBB.pl?action=downloadfile;file=pX330a_Zhang_vector_by_Addgene.gb (16 KB | )
ясно, спасибо, надо будет попробовать, когда выйдет 1.14. Кажется, там намечается много полезного!
Ольга,
вот архив с ab1 файлами, которые я как раз использую: https://yadi.sk/d/EWIHJjgSXBRST
А во вложении последовательность плазмиды, которую я проверяю.
 https://forum.ugene.net/forum/YaBB.pl?action=downloadfile;file=pX330a_Zhang_vector_by_Addgene.gb (16 KB | )
https://forum.ugene.net/forum/YaBB.pl?action=downloadfile;file=pX330a_Zhang_vector_by_Addgene.gb (16 KB | )